はじめに
これまでも何回か扱ってきた決定木ですが、今回は決定木を可視化して解釈する部分を解説します。決定木分析を使うのは、結果の説明を求められるときが多いかと思います。そこで、可視化した結果をどのように解釈して説明すればよいのか、を考えることにしましょう。
過去の決定木の記事もよければ参考にして下さい。
ランダムフォレストと合わせて、決定木の可視化や重要度を解説した記事はこちらです。
サンプルデータ
まずはサンプルデータを用意しましょう。今回は前回の投稿と同様に、sklearnにあらかじめ用意されている「腫瘍細胞に関するデータ」を扱うようにしましょう。
#ライブラリの読み込み import pandas as pd from sklearn.tree import DecisionTreeClassifier, plot_tree from sklearn.model_selection import train_test_split import matplotlib.pyplot as plt #データの読み込み from sklearn.datasets import load_breast_cancer cancer = load_breast_cancer() #データの読み込み from sklearn.datasets import load_breast_cancer cancer = load_breast_cancer() cancer
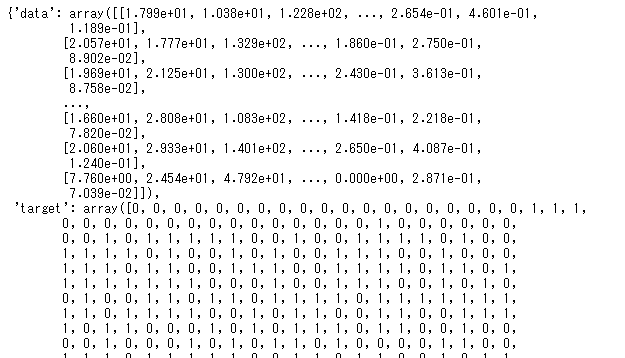
読み込んだcancerは辞書型になっていることがわかります。そこで、どんなkeyがあるのか調べてみましょう。
cancer.keys()
![]()
「data」が説明変数で「target」が目的変数っぽいです。さらにfeature_namesが説明変数のカラム名で、「target_name」が目的変数の値になっているようですね。
決定木
決定木による分類
説明変数と目的変数の準備からはじめて一気にモデルの学習まで進めてしまいましょう。
# 説明変数と目的変数 X=cancer.data y=cancer.target # 訓練データ(train)と検証データ(test)にわける X_train,X_test,y_train,y_test=train_test_split(X,y,test_size=0.3,random_state=1) # モデルの作成 clf = DecisionTreeClassifier(max_depth=3) # モデルの学習 clf.fit(X_train, y_train) # 評価 print(clf.score(X_train,y_train)) print(clf.score(X_test,y_test))
- 学習データで正解率:97.5%
- テストデータで正解率:91.2%
という結果となりました。
決定木の可視化と解釈
次にこの決定木による分類の過程を可視化してみましょう。可視化のコードはたった2行です。
# 可視化 fig = plt.figure(figsize=(20,8)) plot_tree(clf, feature_names=cancer.feature_names, proportion=True, fontsize=10.5, filled=True)
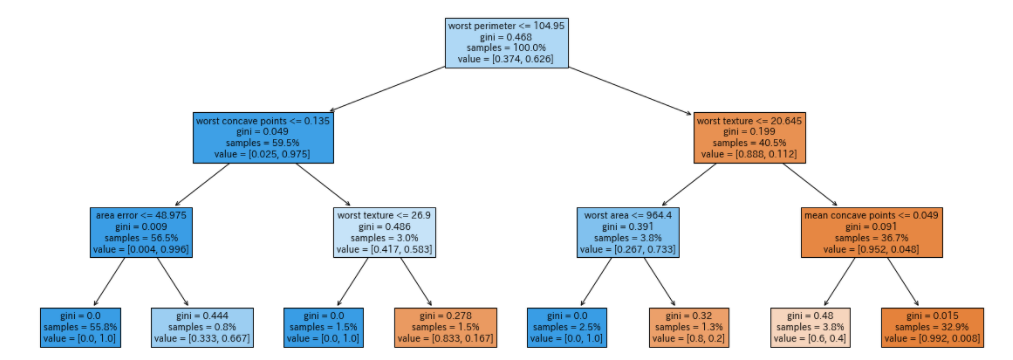
これだけで可視化ができるってすごいですよね。この分類の過程を見ていくことにしましょう。

まずスタートの資格の中をみてみましょう。一番上には、第一段階の分類をするための条件が表示されています。ここでは、「worst perimeter<=104.95」がその条件になります。
2行目はこの条件が何を基に決められたかを表しています。ここでは、「gini」と表示されているので、「ジニ不純度」が小さくなるように決められています。これは、モデル作成時に「criterion=’gini’」のように明示的に指定することもできます。(defaultでジニ不純度となります。)ここで「criterion=’entropy’」とするとエントロピーとなります。
3行目はこのノードで全体の何%のサンプルがあるかを示しています。スタートのノードなので、ここでは100%となっています。最後の「value」は現段階での構成比を表しています。目的変数0のものが37.4%、1のものが63%あります。
このようの記載された条件が「True」のときに左に進み、「False」のときに右に進みます。
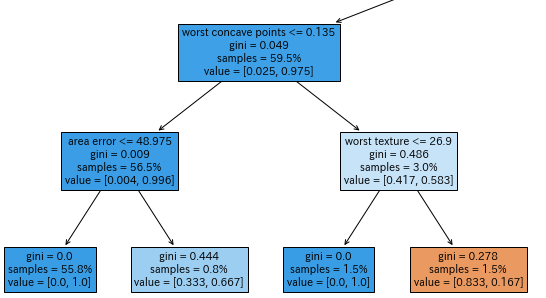
次に、1つめの条件で「True」だった場合、つまり左に進んだ時をみてみましょう。実は各ノードの色にも意味があります。targetの値で0が多いノードは青系の色、1が多いノードはオレンジ系の色になります。以下は、順に同じように解釈していくことができます。
1つ注意点ですが、targetは今回は0/1で分類しているので、上記のとおりですが、これにラベルを付けた場合は「ラベルのアルファベット順」に並び替えられる点に注意が必要がです。今回はtargetの値が0:malignant(悪性)、1:benign(良性)です。そのためアルファベット順に並べると、これが反転します。
この動きを確認しておきましょう。
targetにラベルをつけて同じようにモデルを作成して正解率をみてみましょう。
# targetにラベルをつける y_name=[cancer.target_names[i] for i in y] # 訓練データ(train)と検証データ(test)にわける X_train,X_test,y_train,y_test=train_test_split(X,y_name,test_size=0.3,random_state=1) # モデルの作成 model = DecisionTreeClassifier(max_depth=3) # モデルの学習 model.fit(X_train, y_train) # 評価 print(model.score(X_train,y_train)) print(model.score(X_test,y_test))
![]()
(当たり前ですが・・・)ここまで全く同じ結果が得られています。これを可視化してみましょう。
fig = plt.figure(figsize=(20,8)) plot_tree(model, feature_names=cancer.feature_names, proportion=True, fontsize=10.5, filled=True)
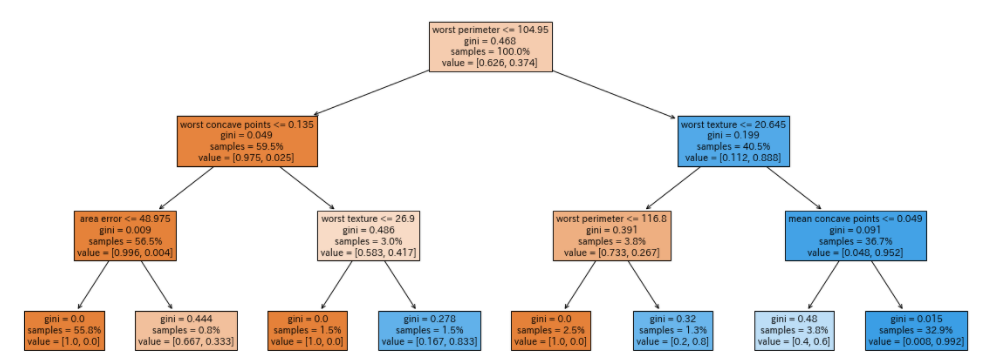
このように条件や分岐などは同じですが、色が反転したのわかりますか?これは、value=[(0の構成比),(1の構成比)]と並んでいたものが、targetにラベルをつけたことによってアルファベット順に並び替えられ、value=[(1の構成比),(0の構成比)]になったためです。
まとめ
いかがでしたか?最後の部分が少しややこしかったかもしれませんね。樹形図は色にも意味があることを知っておくと、パッと見でどのようなに分類が進んでいるかがわかります。実際にコードをいろいろ変更して試してみると、理解が深まると思います。



コメント