はじめに
今回は実際に東京都のWebページで公開されている、コロナウィルス感染者数の推移を可視化してみます。いままでpandasを使っていろんな処理をしてきましたが、今回は、「pandasを使わずに処理」と「pandasを使って処理」の両方を試してみましょう。
データの取得
まずはデータを取得します。東京都のWebページのトップページに「新型コロナウィルス感染症対策サイト(COVID-19)」へのリンクがあります。このリンクを踏んで、サイトに飛ぶと、「陽性患者の属性」というエリアがあります。このエリア内の「オープンデータを入手」をクリックしてデータを取得しましょう。
pandasを使わないで処理
では、「covid.py」というファイルを作って処理していきましょう。csvファイルを読み込むのでcsvライブラリのインポート、感染者数の推移をグラフ化するのでmatplotlibライブラリのインポートをしておきましょう。
# ライブラリのインポート
import csv
import matplotlib.pyplot as plt次に取得したファイルを開きます。With文を使うとClose処理を自動でやってくれるのでしたね。まずはうまく読み込めているかを確認してみましょう。
with open ('20200504_patients.csv',encoding='utf-8') as fh:
reader=csv.reader(fh)
# 1行目を飛ばす
next(reader)
for row in list(reader)[0:5]:
print(row)これを実行して、次のように表示されたら成功です。

csv.readerはリストではないため、インデックスをサポートしていません。そこで、リストに変換して始めの5行のみをfor文で表示しています。うまく表示できているので、この部分はリストではなく_csv.readerのまま扱うことにしましょう。次に日付ごとの感染者の集計をしてみましょう。データは1レコード1感染者となっているので、4列目の日付の数を数えればよいですね。つぎのようにします。
(略)
# 日付ごとに集計
datedic={}
for row in reader:
date=row[4]
if date in datedic.keys():
datedic2026/01/28+=1
else:
datedic2026/01/28=1
for key,value in datedic.items():
print(key,value)これを実行すると次のように表示されます。うまく表示されていたら、for key,value in datedic.items(): ・・・の部分は確認用のコードなので削除してください。
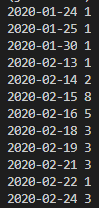
日付ごとの感染者数がわかりました。次にこれをグラフで表すことを考えましょう。いま、データはdatedicという辞書型で管理していますが、辞書型ではデータの並び順は保証されません。そのため、リスト型で管理するようにしましょう。
# リスト型に変換
datelist=[]
for key,value in datedic.items():
datelist.append((key,value))
print(datelist)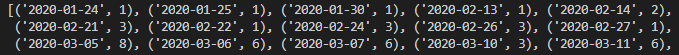
これでリスト型(タプルのリスト)に変換できました。これを日付順に並び変えるようにしましょう。
detalist=sorted(datalist,key=lambda x:x[0])これはlambda式を使えば簡単ですね。sortedは並び替えた結果を返す関数なので、並び替えた結果が欲しければ代入する必要があります。lambda式の部分は、keyのところにlambda式で抽出した日付を指定すればOKですね。
ここまでできたら、描画のための準備です。x軸(日付)とy軸(感染者数)のデータを作っていきましょう。それぞれdates, valuesというリストにしましょう。
dates=[]
values=[]
for date,value in datalist:
dates.append(date)
values.append(value)これを次のようにmatplotlibで描画します。
plt.figure(figsize=(10,10))
plt.bar(dates,values)
plt.xticks(rotation=90)
plt.show()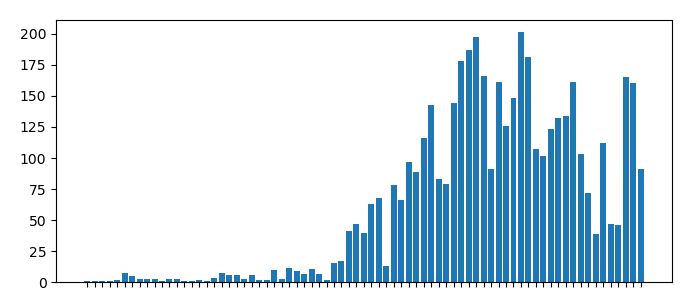
軸ラベルの調整はしていませんが、一応、日付ごとの感染者数をプロットすることができました。次に、pandasを使った処理をしてみましょう。
pandasを使って処理
前回の可視化のsummaryをやりましたね。
これに従ってまずはライブラリのインポートをしましょう。深く考えず、以下のライブラリのインポートと初期設定をワンセットとします。
# ライブラリのインポート
import numpy as np
import pandas as pd
import pandas_profiling
import matplotlib.pyplot as plt
import seaborn as sns
import japanize_matplotlib
# 初期設定
sns.set_style("whitegrid", {'grid.linestyle': '--'})
%matplotlib inline次にデータの読み込みです。
# データの読み込み
_data=pd.read_csv('20200504_patients.csv')
# カラム名の編集
_data.columns=['No', '公共団体コード', '都道府県名', '市区町村名', '公表_年月日', '曜日', '発症_年月日',
'患者_居住地', '患者_年代', '患者_性別', '患者_属性', '患者_状態', '患者_症状', '患者_渡航歴の有無フラグ',
'備考', '退院済フラグ']
# 必要なカラムのみ抽出
data=_data[['No', '都道府県名','公表_年月日', '曜日','患者_居住地', '患者_年代', '患者_性別']]
data.head()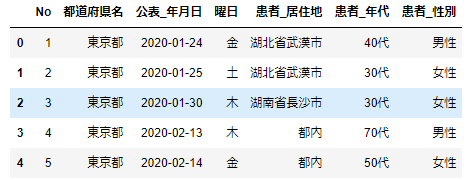
次に日別の感染者数を計算しましょう。これはgroupbyを使うと簡単にできます。
# 日別の感染者数の算出
data_sum=data.groupby('公表_年月日').agg({'No':'count'}).reset_index()
data_sum.columns=['公表_年月日','感染者数']
data_sum.sort_values(by='公表_年月日',inplace=True)グラフ化したときにX軸の日付がきれいに表示されるように少し修正をしておきましょう。
# datetimeライブラリのインポート
import datetime as dt
# フォーマットの変更
data_sum['公表_年月日']=pd.to_datetime(data_sum['公表_年月日'])
data_sum['公表日']=data_sum['公表_年月日'].dt.strftime('%m-%d')
data_sum.head()まず、「公表_年月日」をdatetime型に変換して、そのあとにフォーマットを変更してあらたに「公表日」という列を作っています。
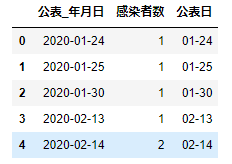
これを可視化してみましょう。
plt.figure(figsize=(15,6))
plt.bar(data_sum['公表日'],data_sum['感染者数'])
plt.ylabel('感染者数(人)')
plt.title('東京都の感染者数の推移',fontsize=18)
plt.xticks(rotation=90)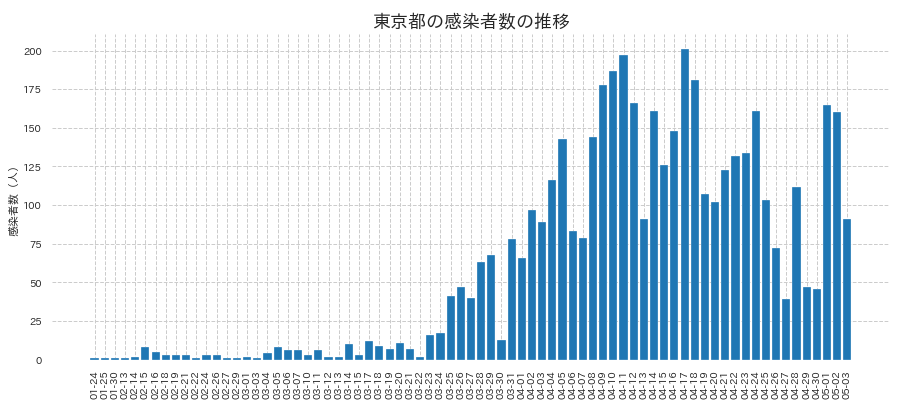
きれいに描けましたね。4/29,30で1日の感染者数が50人を下回って減ったかのように見えましたが、翌日の5/1,2と連続で150人越えとなりまだまだ沈静化してませんね。
まとめ
今回は実データを使って可視化をしてみました。やはりpandasを使った方が圧倒的に楽ですね。今回は時系列にプロットしただけでしたが、次回は少しデータを加工してみていきましょう。


コメント